Virologische Diagnostik bei Hepatitis C und Ihre Bedeutung
Mit Ausnahme der Suche nach einer sehr frühen Hepatitis C Virus (HCV) Infektion wird die Diagnose der HCV Infektion durch Nachweis von Antikörpern gegen Hepatitis C Virus durchgeführt. Die Bestätigung mittels rekombinantem Immunoblot (RIBA) ist in den Hintergrund getreten. An erster Stelle steht hier der Nachweis von HCV-RNA als Hinweis auf eine chronisch aktive Replikation. Erst falls dieser Nachweis nicht gelingt, kann eine serologische Bestätigungsdiagnostik angeschlossen werden, um ggf. eine Kreuzreaktion mit anderen Viren aus der Gruppe der Flaviviridae (z.B. nach Impfung gegen FSME, Gelbfieber) oder eine unspezifische Reaktivität auszuschließen(Cornberg et al., 2011).
Vor Therapiebeginn
Die HCV-Genotypisierung ist entscheidend für die Therapieauswahl (Sarrazin et al., 2015). Derzeit stehen für bestimmte Genotypen inzwischen gut erprobte Kombinationen mit genotyp-spezifischem Spektrum zur Verfügung. Insgesamt hat somit die Bedeutung der HCV-Genotypisierung zugenommen. Bei der Entwicklung neuer Medikamente wird allerdings immer mehr auf eine pangenotypische Wirksamkeit geachtet(Feld et al., 2015)
Probleme bei Genotypisierung
Die HCV-Genotypisierung ist eine seit Jahrzehnten in vielen Laboren durchgeführte Standardmethode mit diversen kommerziell erhältlichen Testsystem und es existieren durchRingversuche eigentlich entsprechende qualitätssichernde Maßnahmen. Dennoch zeigen sich mit der Verwendung der direkt aktiven antiviralen (DAA) Therapie teilweise systematische Probleme. Um eine hohe Rate erfolgreicher Genotypisierungen zu realisieren, wurden häufig hoch konservierte Genbereiche (z.B. 5‘-UTR) zur Bestimmung verwendet. Eine Einteilung in die Genotypen 1 bis 4 gelingt häufig, eine Klassifizierung der Zugehörigkeit zu einem Subtyp 1a und 1b ist hier nur bedingt mit ausreichender Sicherheit möglich. Eine Entscheidung zwischen Clade I und Clade II des Genotyps 1a ist mit diesen Systemen vollkommen ausgeschlossen. Häufig sind diese Systeme auch anfällig auf Fehlbestimmungen der Genotypen 5 und 6, wobei diese häufig zu falsch klassifizierten Genotyp 1 Ergebnissen führen (Chueca et al., 2016).
Eine weitere Hürde zur korrekten Genotypisierung ist die Möglichkeit von rekombinanten HCV-Stämmen. Ein bekanntes Beispiel ist die sogenannte St. Petersburg-Variante, eine Rekombination aus einem Genotyp 2k und einem Genotyp 1b Virus (Schuermans et al., 2016). Von den meisten Systemen als Genotyp 2 erkannt, ist diese in den für die Behandlung mit DAAs relevanten Genbereichen aber dem Genotyp 1b zugehörig und sollte somit auch wie dieser Genotyp behandelt werden. Auch Mischinfektionen mit verschiedenen HCV-Genotypen erschweren die Genotypisierung, wobei die Häufigkeit in der Literatur, abhängig von der verwendeten Methode und der untersuchten Population zwischen 2% und 10% angegben wird(Giannini et al., 1999; Viazov et al., n.d.; Walker et al., 2016).
Viruslast
Die Bestimmung der HCV-Last in Serum oder Plasma mittels Nukleinsäure-Amplifikations-Technik (NAT) ist ein fester Bestandteil der Untersuchungen vor Therapiebeginn. Obwohl ein von der WHO freigegebener Standard inzwischen schon in der 5. Generation existiert (“EXPERT COMMITTEE ON BIOLOGICAL STANDARDIZATION Geneva, 12 to 16 October 2015,” n.d.), gibt es vor allem im hohen Viruslastbereich, abhängig vom Genotyp, deutliche Unterschiede bis Faktor 10 zwischen den Verfahren einzelner Hersteller dieser Testverfahren (Abbildung 1). Auch wenn die Reproduzierbarkeit der Messwerte in diesem Bereich sehr gut ist, sind Schwankungen um den Faktor zwei bis drei in Einzelfällen bei wiederholter Messung derselben Probe zu beobachten. Weiter kompliziert wird dies durch individuelle Viruslastverläufe, die innerhalb weniger Wochen um mehrere Faktor 10-Stufen schwanken. Eine Therapieentscheidung und eine Therapieverkürzung sollte also auf jeden Fall deutlich mehrere Faktoren als eine punktuelle Viruslastbestimmung beinhalten.
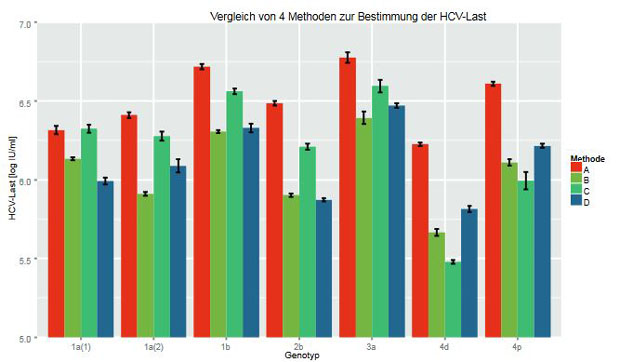
Abbildung
1: Logarithmische Darstellung der Viruslastergebnisse 6 verschiedener
Proben mit 5 unter - schiedlichen (Sub-)Genotypen in fünffach-Ansatz mit
4 verschiedenen Testverfahren
Unter Therapie
Mit den neuen DAA-Therapien erfolgt derzeit keine „response-guided-therapy“ mehr, d.h. die Therapie wird nicht mehr der Kinetik des Virusabfalls angepasst. Dennoch untersuchen derzeit mehrere Arbeitsgruppen dieses Thema, vor allem in Bezug auf Therapie-Verkürzung. Unabhängig davon ist der Verlauf der Viruslast unter Therapie sowohl für den Behandler als auch für den Patienten ein wichtiger Parameter den Erfolg der Therapie zu beurteilen. Zu Verwirrung führt hier, daß bedingt durch die inzwischen immer empfindlicheren Testverfahren, die einen rein qualitativen Nachweis von HCV-RNA obsolet machen, zu Therapie-Ende in Einzelfällen eine geringe Menge von HCV-RNA nachgewiesen wird (<25 IU/ml). Die Bedeutung dieser Nachweise ist derzeit ebenso wie die Ursachen, völlig unklar. Es kann aber in keiner Hinsicht auf einen fehlenden Therapieerfolg geschlossen werden oder daraus eine Indikation zur Therapieverlängerung hergeleitet werden (Sidharthan et al., 2015).
Nach Therapie
Spätestens 12 Wochen nach Beendigung der Therapie sollte HCV auch mit empfindlichsten Testverfahren nicht mehr nachweisbar sein. Kann auch 24 Wochen nach Therapieende kein Virus mehr nachgewiesen werden, gilt die Hepatitis C Infektion als sicher ausgeheilt. Bei einem erneutem Nachweis von HCV-RNA sollte man trotz immer wieder auftauchender Berichte von einem sogenannten „late relaps“ an eine Re-Infektion denken, inbesondere bei weiterbestehendem Infektionsrisiko. Eine klassische Genotypisierung ist zur Klärung der Frage Re-Infektion oder relapse nicht ausreichend. Da häufig eine Reinfektion mit dem gleichen Genotyp beobachtet wird, führt eigentlich nur eine Sequenzanalyse und diese teilweise nur unter Zuhilfenahme einer phylogenetischen Rekonstruktion („Virus-Stammbaum“) zu einer Klärung, ob dasselbe Virus oder ein neuer Virusstamm nachweisbar ist.
Resistenztest vor Therapie
Aufgrund der sehr hohen Erfolgsraten der DAA-Therapie stellt sich immer wieder die Frage nach der Notwendigkeit einer Resistenztestung. Die HCV-Therapie kann trotz Nachweis einer Resistenz assozierten Variante (RAV) mit entsprechenden Therapie-relevanten Mutationen erfolgreich sein. Dies liegt in erster Linie an der Tatsache, dass immer eine Kombinationstherapie durchgeführt wird. Die Wahrscheinlichkeit, dass Resistenzen gegen mehrere Medikamente der Kombination vorliegen, ist sehr unwahrscheinlich. Die Häufigkeit einer Resistenz ist zudem stark vom Medikament und somit dem relevanten Genbereich abhängig. Mutationen im NS3 mit relevanter Resistenz gegen Protease-Inhibitoren sind mit einer Ausnahme grundsätzlich selten. Die Ausnahme: Beim Genotyp 1a schwankt die Prävalenz für die Clade I typische Mutation Q80K regional stark (3% bis 50 %) (Ehret et al., 2014). Die Wirksamkeit von Simeprevir ist bei diesen Viren deutlich eingeschränkt und Alternativen sollten bevorzugt werden.
Im NS5A ist die Situation deutlich unübersichtlicher. Mutationen haben oft nur in einem bestimmten Genotyp-Zusammenhang eine Bedeutung und sind bei anderen Genotypen als natürlich vorkommende Varianten nicht mit einem schlechteren Therapieansprechen verbunden. Mutationen im NS5A treten mit einer Häufigkeit von bis zu 30% oder sogar höher auf (Patiño-Galindo et al., 2016). Im NS5B muss zwischen dem nukleotidischem Inhibitor Sofosbuvir und den nicht-nukleosidischen Inhibitoren (z.B. Dasabuvir) unterschieden werden. Während Resistenzen gegen Sofosbuvir als Raritäten gelten, sind Resistenz-assoziierte Mutationen gegen Dasabuvir regelmäßig zu beobachten. Bei Erst-Therapie ist der Einfluss dieser Mutationen nach derzeitiger Studienlage aber gering. Ob die bisher dafür durchgeführten Studien geeignet sind den Einfluss der Mutationen auf den Therapieerfolg zu belegen, ist aber tatsächlich schwierig zu beurteilen. So werden oft alle Resistenz-assoziierten Mutationen aufgezählt, unabhängig davon, ob sie für die Folgetherapie eine Rolle spielen oder nicht. So ist eine Resistenz gegen einen Protease-Inhibitor tatsächlich unerheblich für eine Therapie mit einem NS5A- und einem NS5B-Inhibitor und bestätigt nur die erwartete fehlende Kreuzresistenz zwischen den einzelnen Substanzklassen. Falls die Mutationen aber in den Studien gezielt einzeln ausgewertet werden, zeigt sich die unterschiedliche Bedeutung einzelner Mutationen. Diese Daten muss man aber nicht selbst einzeln Zusammentragen. So wurden für den geno2pheno-Service diese Daten zusammengetragen und auch publiziert (Kalaghatgi et al., 2016).
Nach DAA-Versagen
Die Resistenztestung nach Therapieversagen zur Planung einer Folgetherapie hat sich inzwischen durchgesetzt (Sarrazin, 2015). Hier sollen weiter erfolglose Therapieversuche vermieden werden und die Therapieauswahl vereinfacht werden. Moderne Techniken zur Sequenzanalyse („next generation Sequencing“, NGS) ermöglichen es, größere Stücke des HCV-Genoms in einem kürzeren Zeitraum zu untersuchen als mit der klassischen Sequenzierung (Wei et al., 2016). So können die Genbereiche NS3, NS5A und NS5B gleichzeitig auch auf das Vorhandensein von minoren Populationen untersucht werden (Quer et al., 2015). Dabei handelt es sich um eine Resistenz-assoziierten Variante, die nur in kleiner Menge (Populationsanteil) neben der empfindlichen Variante nachweisbar ist. Im Unterschied zu HIV gibt es keinen Latenz-Ort für das Hepatitis C Virus und somit auch kein Archiv für resistente Varianten. Mutationen können somit im Verlauf der Zeit tatsächlich verschwinden. Auch wenn es sich teilweise um viele Monate handelt, bis eine Resistenz-assoziierte Variante verschwunden ist, ermöglichen die NGS-Verfahren eine Überprüfung, ob eine Mutation noch in relevanter Menge vorhanden ist. Als Nebenbefund dieser Verfahren erhält man eine sehr solide Bestimmung des Genotyps aus den für die Behandlung relevanten Genbereichen.
Chueca, N., Rivadulla, I., Lovatti, R., Reina, G., Blanco, A., Fernandez-Caballero, J.A., Cardeñoso, L., Rodriguez-Granjer, J., Fernandez-Alonso, M., Aguilera, A., Alvarez, M., Galán, J.C., García, F., 2016. Using NS5B Sequencing for Hepatitis C Virus Genotyping Reveals Discordances with Commercial Platforms. PLOS ONE 11, e0153754. doi:10.1371/journal.pone.0153754
Cornberg, M., Protzer, U., Petersen, J., Wedemeyer, H., Berg, T., Jilg, W., Erhardt, A., Wirth, S., Sarrazin, C., Dollinger10, M.M., 2011. Aktualisierung der S 3-Leitlinie zur Prophylaxe, Diagnostik und Therapie der Hepatitis-B-Virusinfektion. Z Gastroenterol 49, 871–930.
Ehret, R., Neifer, S., Walter, H., Baumgarten, A., Obermeier, M., 2014. Appearance of NS3 Q80K mutation in HCV genotype 1a mono- or HIV/HCV co-infected patients in a Berlin laboratory. J. Int. AIDS Soc. 17. doi:10.7448/IAS.17.4.19741
EXPERT COMMITTEE ON BIOLOGICAL STANDARDIZATION Geneva, 12 to 16 October 2015 [WWW Document], n.d. URL http://www.who.int/biologicals/expert_committee/BS2262_Establishment_HCV-RNA_5th_WHO_IS.pdf (accessed 9.6.16).
Feld, J.J., Jacobson, I.M., Hézode, C., Asselah, T., Ruane, P.J., Gruener, N., Abergel, A., Mangia, A., Lai, C.-L., Chan, H.L.Y., Mazzotta, F., Moreno, C., Yoshida, E., Shafran, S.D., Towner, W.J., Tran, T.T., McNally, J., Osinusi, A., Svarovskaia, E., Zhu, Y., Brainard, D.M., McHutchison, J.G., Agarwal, K., Zeuzem, S., 2015. Sofosbuvir and Velpatasvir for HCV Genotype 1, 2, 4, 5, and 6 Infection. N. Engl. J. Med. 373, 2599–2607. doi:10.1056/NEJMoa1512610
Giannini, C., Giannelli, F., Monti, M., Careccia, G., Marrocchi, M.E., Laffi, G., Gentilini, P., Zignego, A.L., 1999. Prevalence of mixed infection by different hepatitis C virus genotypes in patients with hepatitis C virus-related chronic liver disease. J. Lab. Clin. Med. 134, 68–73.
Kalaghatgi, P., Sikorski, A.M., Knops, E., Rupp, D., Sierra, S., Heger, E., Neumann-Fraune, M., Beggel, B., Walker, A., Timm, J., Walter, H., Obermeier, M., Kaiser, R., Bartenschlager, R., Lengauer, T., 2016. Geno2pheno[HCV] – A Web-based Interpretation System to Support Hepatitis C Treatment Decisions in the Era of Direct-Acting Antiviral Agents. PLOS ONE 11, e0155869. doi:10.1371/journal.pone.0155869
Patiño-Galindo, J.Á., Salvatierra, K., González-Candelas, F., López-Labrador, F.X., 2016. Comprehensive screening for naturally-occurring Hepatitis C virus resistance to direct-acting antivirals in the NS3, NS5A and NS5B genes in worldwide isolates from viral genotypes 1-6. Antimicrob. Agents Chemother. AAC.02776-15. doi:10.1128/AAC.02776-15
Quer, J., Gregori, J., Rodríguez-Frias, F., Buti, M., Madejon, A., Perez-del-Pulgar, S., Garcia-Cehic, D., Casillas, R., Blasi, M., Homs, M., Tabernero, D., Alvarez-Tejado, M., Muñoz, J.M., Cubero, M., Caballero, A., delCampo, J.A., Domingo, E., Belmonte, I., Nieto, L., Lens, S., Muñoz-de-Rueda, P., Sanz-Cameno, P., Sauleda, S., Bes, M., Gomez, J., Briones, C., Perales, C., Sheldon, J., Castells, L., Viladomiu, L., Salmeron, J., Ruiz-Extremera, A., Quiles-Pérez, R., Moreno-Otero, R., López-Rodríguez, R., Allende, H., Romero-Gómez, M., Guardia, J., Esteban, R., Garcia-Samaniego, J., Forns, X., Esteban, J.I., 2015. High-Resolution Hepatitis C Virus Subtyping Using NS5B Deep Sequencing and Phylogeny, an Alternative to Current Methods. J. Clin. Microbiol. 53, 219–226. doi:10.1128/JCM.02093-14
Sarrazin, C., 2015. The importance of resistance to direct antiviral drugs in HCV infection in clinical practice. J. Hepatol. doi:10.1016/j.jhep.2015.09.011
Sarrazin, C., Berg, T., Buggisch, P., Dollinger, M., Hinrichsen, H., Hofer, H., Hüppe, D., Manns, M., Mauss, S., Petersen, J., Simon, K.-G., van Thiel, I., Wedemeyer, H., Zeuzem, S., 2015. Aktuelle Empfehlung zur Therapie der chronischen Hepatitis C. Z. Für Gastroenterol. 53, 320–334. doi:10.1055/s-0034-1399322
Schuermans, W., Orlent, H., Desombere, I., Descheemaeker, P., Van Vlierberghe, H., Geerts, A., Verhelst, X., Reynders, M., Padalko, E., 2016. Heads or Tails: Genotyping of Hepatitis C Virus Concerning the 2k/1b Circulating Recombinant Form. Int. J. Mol. Sci. 17, 1384. doi:10.3390/ijms17091384
Sidharthan, S., Kohli, A., Sims, Z., Nelson, A., Osinusi, A., Masur, H., Kottilil, S., 2015. Utility of Hepatitis C Viral Load Monitoring on Direct-Acting Antiviral Therapy. Clin. Infect. Dis. 60, 1743–1751. doi:10.1093/cid/civ170
Viazov, S., Widell, A., Nordenfelt, E., n.d. Mixed Infection with Two Types of Hepatitis C Virus Is Probably a Rare Event. Infection 28, 21–25. doi:10.1007/s150100050005
Walker, M.R., Li, H., Teutsch, S., Betz-Stablein, B., Luciani, F., Lloyd, A.R., Bull, R.A., 2016. Incident Hepatitis C Virus Genotype Distribution and Multiple Infection in Australian Prisons. J. Clin. Microbiol. 54, 1855–1861. doi:10.1128/JCM.00287-16
Wei, B., Kang, J., Kibukawa, M., Chen, L., Qiu, P., Lahser, F., Marton, M., Levitan, D., 2016. Development and Validation of a Template-Independent Next-Generation Sequencing Assay for Detecting Low-Level Resistance-Associated Variants of Hepatitis C Virus. J. Mol. Diagn. 18, 643–656.