R. S. Ross*, S. Viazov, S. Salloum, M. Roggendorf, Essen
Analytische Kenndaten und Einsatzmöglichkeiten des ARCHITECT-HCV-Ag-Assays
Die gegenwärtige Diagnostik von HCV-Infektionen basiert auf der Detektion von anti-HCV-Antikörpern mit Immunoassays der dritten Generation und erfordert die Bestätigung eines initial als reaktiv erhobenen Befunds durch einen HCV-RNA-Nachweis, der prinzipiell eine Unterscheidung zwischen einer abgelaufenen und einer unverändert bestehenden und damit replikativen chronischen Infektion ermöglicht.1, 2
Da allerdings Methoden zur Nukleinsäureamplifikation noch immer vergleichsweise kostspielig sind und es zu ihrer Durchführung sowohl eines entsprechend ausgestatteten Laboratoriums als auch hinreichend geschulten Personals bedarf, wurde in den zurückliegenden rund zehn Jahren immer wieder der Nachweis des HCV-Core-Antigens als Alternative zur HCV-RNA-Bestimmung propagiert.3-5 Sämtliche vorgeschlagenen Testformate konnten sich jedoch bislang nicht durchsetzen, wobei ihre unzureichende analytische Sensitivität als einer der hauptsächlichen Kritikpunkte galt.
Der von Abbott Diagnostics im vergangenen Jahr auf dem diagnostischen Markt etablierte ARCHITECT-HCV-Ag-Assay schickt sich nun an, die Unzulänglichkeiten seiner diversen Vorgänger zu überwinden. Als Nationalem Referenzzentrum für HCV bot sich uns die Möglichkeit, diesen neu entwickelten Testkit zur Bestimmung des gesamten HCV-Core-Antigens noch vor der Markteinführung ausführlich zu evaluieren. Die Untersuchungen zur Bestimmung der rein analytischen Kenngrößen orientierten sich an dem einschlägigen und ursprünglich für klinisch-chemische Analysatoren konzipierten ECCLS-Protokoll.6 Die diagnostischen Kennwerte wurden anhand von Proben mehrerer Hundert chronisch HCV-Infizierter ermittelt.7
Analytische Kenngrößen
Die Untersuchung von 100 anti-HCV-negativen Seren, die von gesunden Probanden stammten, erbrachte eine Spezifität von 100% (95% Konfidenzintervall: 96,4-100%). Die Reproduzierbarkeit innerhalb eines Laufes betrug konzentrationsabhängig 3,6-8,0% und belief sich im Inter-Assay-Vergleich auf 4,7-9,5%. Mit Ausnahme von HCV-Subtyp-2-Isolaten wurden alle viralen Varianten mit 95%iger Wahrscheinlichkeit in Konzentrationen von unter 10 fmol HCV-Core-Antigen/l erkannt.
Die diagnostische Sensitivität des Tests ermittelten wir durch die Untersuchung dreier kommerziell erhältlicher Serokonversionspanels. Dabei konnte durch den Core-Antigen-Nachweis das diagnostische (antikörper-negative) Fenster um 4-16 Tage verkürzt werden, womit sich der Assay als ebenso sensitiv wie die HCV-RNA-Bestimmung erwies.
Der lineare Bereich des ARCHITECT-HCV-Ag-Tests, in dem verlässliche quantitative Werte ermittelt werden konnten, bewegte sich zwischen 10 und 20.000 fmol/l.
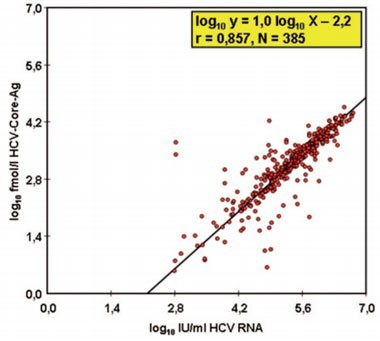
Abb. 1: Punktdiagramm, das die Korrelation von log 10 HCV-Core-Antigen- und log 10 HCV-RNA-Konzentrationen zeigt, wie sie sich bei der vergleichenden Untersuchung von 385 Patientenproben mittels des ARCHITECT-HCV-Ag- und des VERSANT HCV RNA 3.0 Assays ergaben. Der Korrelationskoeffizient und die Regressionsgerade wurden durch Anwendung der nicht-parametrischen Methode nach Passing und Bablock 11 ermittelt (verändert nach 7 )
Die HCV-Core-Antigen- und HCV-RNA-Konzentrationen waren bei der vergleichenden Untersuchung von 385 Proben miteinander korreliert (Abb. 1), jedoch ließ die Regressionsgerade eine signifikante Abweichung von der Winkelhalbierenden erkennen: log10 HCV-Core-Antigen (fmol/l) = 1,0 x log10 HCV RNA (IU/ml) – 2,2 (95% Konfidenzintervalle für die Steigung und den Achsenabschnitt: 0,966 – 1,026 und – 2,343 - - 2,012). Separate Auswertungen für die HCV-Subtypen 1a und 1b sowie die Genotypen 2 und 3 führten zu ähnlichen Regressionsgeraden mit Achsenabschnitten, die zwischen – 2,085 (HCV-Subtyp 1b) und – 2,571 (HCV-Genotyp 3) schwankten. Ein fmol HCV-Core-Antigen/l entsprach somit rechnerisch zwischen 135 IU (HCV-Subtyp 1a) und 257 IU (HCV-Genotyp 3) HCV RNA/ml.
Diagnostische Kenngrößen
Neunzig Seren, die von HCV-infizierten virämischen Hämodialyse-Patienten stammten, wurden sowohl mit dem ARCHITECT-HCV-Ag-Assay als auch mit einem seiner Vorgänger, dem trak-C-Test von Johnson & Johnson, Ortho Clinical Diagnostics, untersucht. Achtzig der Proben waren in beiden Testformaten reaktiv, jedoch erkannte der ARCHITECT-Assay auch zehn Materialien, deren mittlere HCV-RNA-Konzentration nur 1.152 IU/ml betrug und die daher dem Nachweis mit dem analytisch weniger sensitiven trak-C-Kit entgingen.
Produktinformationen zum ARCHITECT-HCV-Ag-Assay der Firma Abbott Diagnostics
Warum HCV Ag?
HCV-Antigen ist vor der Serokonversion, d.h. vor dem Auftreten von HCV-Antikörpern nachweisbar. Nach einer ausgeheilten akuten Infektion können HCV-Antikörper noch vorhanden sein, eine Virämie, bzw. das HCV Ag ist aber nicht mehr nachweisbar.
Klinische Anwendung des HCV Antigen Tests
Screening von Patienten mit hohem Risiko für eine HCV Infektion, z.B.: Dialyse-Patienten, i.v. Drogenabhängige
Reflex-Testung eines Anti-HCV reaktiven Ergebnisses – um eine aktive von einer ausgeheilten Infektion zu unterscheiden
Quantitativer Test als Ergänzung in der Überwachung der antiviralen Therapie
Hohe Spezifität:
99,76% initiale Spezifität
99,98% nach Wiederholung initial reaktiver Proben
Hohe Sensitivität:
Grenzwert: 3 fmol/l
Von 422 RNA-positiven Proben (qualitative PCR, Roche Amplicor) wurden 414 (98,1%) mit dem ARCHITECT HCV Ag Test detektiert und 399 (94,5%) mit einer quantitativen PCR (Roche Amplicor Monitor)
Bei 10 kommerziell erhältlichen Serokonversionspaneln deren erste Probe RNA-positiv war, wurde die erste Probe ebenfalls mit dem ARCHITECT HCV Ag Test detektiert – Verkürzung des serologischen Fensters um 35,8 Tage im Vergleich zur Bestimmung der HCV-Antikörper
* Detaillierte Daten: Morota et al., A fully automated and highly sensitive immunoassay for hepatitis C virus (HCV ) core antigen, P-213, 2007 ISBT Madrid, Spain
Bei insgesamt 38 chronisch mit HCV infizierten
Patienten überwachten wir deren Ansprechen auf eine antivirale
Kombinationsbehandlung mit pegyliertem Interferon a und Ribavirin durch
Bestimmungen der HCV-Core-Antigen- und HCV-RNA-Konzentrationen. Wie aus Abb. 2
exemplarisch ersichtlich ist, stellten sich in allen untersuchten Fällen sehr
ähnliche und daher gleichsam parameterunabhängige Verlaufskurven ein. Die gute
Übereinstimmung der Messwerte auch hinsichtlich der Voraussage des
Therapieerfolgs zeigte sich beispielsweise dann, wenn man auf der Basis des
„2-log-Abfallkriteriums“ zur Behandlungswoche 12 die prädiktiven Werte des
schließlich erreichten dauerhaften
virologischen Ansprechens
berechnete. Sowohl aus dem Verlauf der HCV-Core-Antigen- als auch der
HCV-RNA-Konzentrationen ergaben sich für Patienten, die mit HCV-Isolaten der
Genotypen 1 und 4 infiziert waren, zur Therapiewoche 12 positive und negative
Vorhersagewerte von 45% und 100%. Die „Pendants“ für Personen mit
HCV-Genotyp-2- bzw. -3-Infektionen beliefen sich auf 87,5% und 100%. Diese
Beobachtungen belegten, dass der ARCHITECT-HCV-Ag-Assay prinzipiell ebenso zur
Überwachung einer antiviralen Therapie chronischer HCV-Infektionen geeignet ist
wie die quantitative HCV-RNA-Bestimmung. Angesichts seiner im Vergleich zur
Nukleinsäureamplifikation jedoch noch immer geringeren analytischen
Sensitivität, der insbesondere für Entscheidungen zu bestimmten Zeitpunkten der
Behandlung außerordentliche Bedeutung zukommt,1, 2 und des Fehlens
von Therapiealgorithmen, die in kontrollierten klinischen Studien unter Einsatz
des ARCHITECT-HCV-Ag-Assays entwickelt wurden, erscheint es allerdings
fraglich, ob der Test zukünftig einen Platz im „Therapie-Monitoring“ wird
finden können.
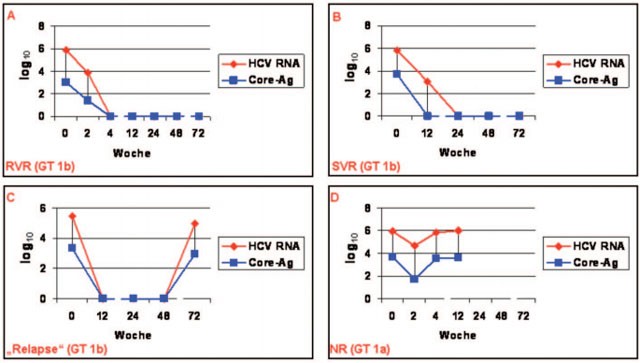
Abb. 2: Kinetiken der HCV-Core-Antigen- und HCV-RNA-Konzentrations-Abnahmen bei vier exemplarisch ausgewählten Patienten, die sich einer Kombinationsbehandlung mit pegyliertem Interferon a und Ribavirin unterzogen. A: Schnelles virologisches Ansprechen (RVR). B: Dauerhaftes virologisches Ansprechen (SVR). C: Patient, der nach Beendigung der antiviralen Behandlung einen „Relapse“ entwickelte. D: Patient, der nicht auf die antivirale Therapie ansprach (NR). GT: HCV-Genotyp (verändert nach 7 )
Eignung zur Überwachung der Replikation in Zellkulturen
Um die Eignung des ARCHITECT-HCV-Ag-Assays zur Überwachung auch der HCV-Replikation in Zellkultursystemen zu prüfen, wurden HuH 7-Zellen eingesetzt, die stabil mit einem AD78-basierten HCV-„full-length-Konstrukt“ transfiziert waren8 und mit unterschiedlichen Konzentrationen von Interferon a bzw. dem Protease-Inhibitor BILN 20619, 10 für 48 Stunden in vitro behandelt wurden. Unter dieser Therapie stellten sich Reduktionen der HCV-Core-Antigen-Konzentrationen auf bis zu 7,9% (Interferon a) und 29,4% (BILN 2061) der jeweiligen Ausgangswerte ein, so dass der ARCHITECT-HCV-Ag-Assay als geeignetes und leicht zu handhabendes Testformat für das „Hochdurchsatz-Monitoring“ der HCV-Replikation in in vitro-Systemen gelten kann.
Zusammenfassung
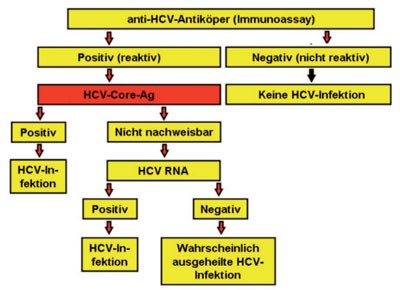 Abb. 3: Möglicher zukünftiger Algorithmus der HCV-Primärdiagnostik unter Einbeziehung des ARCHITECT-HCV-Ag-Assays als Bestätigungstest für initial reaktive anti-HCV-Befunde (verändert nach 12 )
Abb. 3: Möglicher zukünftiger Algorithmus der HCV-Primärdiagnostik unter Einbeziehung des ARCHITECT-HCV-Ag-Assays als Bestätigungstest für initial reaktive anti-HCV-Befunde (verändert nach 12 )
Zusammenfassend erwies sich der ARCHITECT-HCV-Ag-Assay in unserer bereits vor seiner Markteinführung durchgeführten umfangreichen Evaluation als ein spezifisches, reproduzierbares, analytisch und diagnostisch sensitives und mit der HCV-RNA-Konzentration nachvollziehbar korreliertes Verfahren zur quantitativen Bestimmung des HCV-Core-Antigen-Gehalts, das auch klinisch einsetzbar erscheint.7 Nach unserer Überzeugung wird er – sofern es dem Hersteller gelingen sollte, die von uns festgestellte „carry-over-Problematik“ durch geeignete Modifikationen des existierenden Assay-Protokolls zu beseitigen – seine Anwendung wohl vor allem als Bestätigungstest in der Primärdiagnostik der HCV-Infektion finden (Abb. 3) und so in Zukunft möglicherweise bis zu 70% der gegenwärtig in diesem Zusammenhang noch durchgeführten HCV-Nukleinsäurenachweise entbehrlich machen.
1 Sarrazin C, Berg T, Ross RS, Schirmacher P, Wedemeyer H, Neumann U, Schmidt HHJ, Spengler U, Wirth S, Kessler HH, Peck-Radosavljevic, Ferenci P, Vogel W, Moradpour D, Heim M, Cornberg M, Protzer U, Manns MP, Fleig WE, Dollinger MM, Zeuzem S. Update der S3-Leitlinie Prophylaxe, Diagnostik und Therapie der Hepatitis-C-Virus (HCV)-Infektion, AWMF-Register-Nr.: 021/012. Z Gastroenterol 2010; 48: 289-351.
2 National Institute of Health. NIH consensus statement on management of hepatitis C: 2002. NIH Consens State Sci Statements 2002; 19: 1-46.
3 Aoyagy K, Ohue C, Iida K, Kimura T, Tanaka E, Kiyosawa K, Yagi S. Development of a simple and highly sensitive enzyme immunoassay for hepatitis C virus core antigen. J Clin Microbiol 1999; 37: 1802-1808.
4 Bouvier-Alias M, Patel K, Dahari H, Beaucourt S, Larderie P, Blatt L, Hezode C, Picchio G, Dhumeaux D, Neumann AU, McHutchinson JG, Pawlotsky JM. Clinical utility of total HCV core antigen quantification: a new indirect marker of HCV replication. Hepatology 2002; 36: 211-218.
5 Tanaka T, Lau JY, Mizokami M, Orioto E, Tanaka R, Kiyosawa K, Yasui K, Ohta Y, Hasegawa A, Tanaka S, Kahawa H. Simple fluorescent enzyme immunoassay for detection and quantification of hepatitic C viraemia. J Hepatol 1995; 26: 742-745.
6 European Committee for Clinical Laboratory Standards (ECCLS). Guideline for the evaluation of analyzers in clinical chemistry. Document 3, 2. Berlin (Beuth), 1986.
7 Ross RS, Viazov S, Salloum S, Hilgard P, Gerken G,
Roggendorf
M. Analytical performance characteristics and clinical utility of a novel assay
for quantification of total hepatitis C virus core antigen. J Clin Microbiol,
2010; 48: 1161-1168..
8 Oniangue-Ndza C, aus dem Siepen M, Lohmann V, Wiese M, Viazov S,
Roggendorf M. In vitro replicative properties of replicons constructed using
sequence variants
of
the hepatitis C virus strain AD78 that caused a single-source outbreak of
hepatitis C. Virus Res 2009; 142:
1-9.
9 Lamarre D, Anderson PC, Bailey M, Beaulieu P, Bolger G, Bonneau P, Bös M, Cameron DR, Cartier M, Cordingley MG, Faucher AM, Goudreau N, Kawai SH, Kukolj G, Lagacé L, LaPlante SR, Narjes H, Poupart MA, Rancourt J, Sentjens RE, St George R, Simoneau B, Steinmann G, Thibeault D, Tsantrizos YS, Weldon SM, Yong CL, Llinàs-Brunet M. An NS3 protease inhibitor with antiviral effects in humans infected with hepatitis C virus. Nature 2003; 426: 186-189.
10 Hinrichsen H, Benhamou Y, Wedemeyer H, Reiser M, Sentjens RE, Calleja JL, Forns X, Erhardt A, Crönlein J, Chaves RL, Yong CL, Nehmiz G, Steinmann GG. Short-term antiviral efficacy of BILN 2061, a hepatitis C virus serine protease inhibitor, in hepatitis C genotype 1 patients. Gastroenterology 2004; 127: 1347-1355.
11 Passing H, Bablok W. A new biometrical procedure for testing the equality of measurements from two different analytical methods. J Clin Chem Clin Biochem 1983; 21: 709-720.
12 Roggendorf M, Roß RS. Hepatitis C-Virus – Diagnostik und Prävention. In: Doerr HW, Gerlich WH (Hrsg.): Medizinische Virologie. 2. Auflage, Stuttgart, New York (Thieme Verlag), 2009, S. 408-416.